近日,我所生物技术研究部生物分子高效分离与表征研究组(1810组)赵群研究员和张丽华研究员等人与中国科学院精密测量科学技术创新研究院龚洲副研究员合作,提出了利用原位化学交联—质谱技术(in vivo XL-MS),解码细胞中蛋白质动态结构的策略。该策略将AlphaFold2的结构作为先验信息,结合in vivo XL-MS数据与多种结构计算方法评估结构与交联信息的匹配度,重构了细胞内多种蛋白质,尤其是多结构域蛋白质和固有无序蛋白质(intrinsically disordered protein,IDP)的原位动态结构。为深入研究蛋白质在细胞微环境中发挥功能的分子机制提供技术支撑。
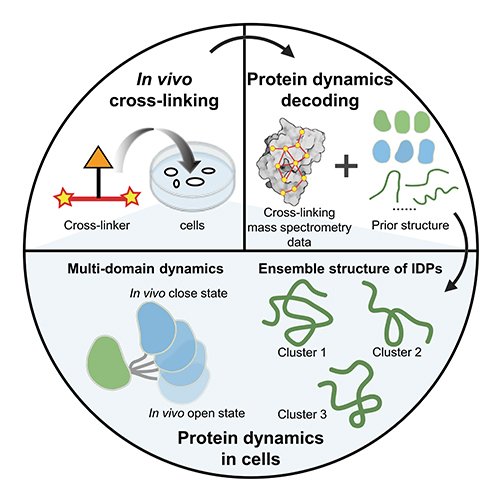
活细胞内蛋白质的原位动态结构对于揭示其生物学功能至关重要。随着深度学习算法助力蛋白质结构预测的发展迭代,AlphaFold2实现了对蛋白质结构的全面预测,然而该方法对柔性区域的结构预测仍面临挑战。近年来,in vivo XL-MS以高通量、高灵敏,且对蛋白质纯度要求低等优势,在解析活细胞内蛋白质的原位动态结构方面展示出重要潜力。张丽华团队一直致力于in vivo XL-MS新技术研究,实现了蛋白质原位构象和相互作用的规模化解析(Anal. Chem.,2020;Anal. Chem.,2022;Anal. Chem.,2022;Anal. Chem.,2022;Anal. Chem.,2023;Angew. Chem. Int. Ed.,2023;Nat. Commun.,2023)。
本工作中,针对多结构域蛋白质,研究团队提出了将结构域作为整体,利用结构域间的XL-MS数据对细胞内蛋白质动态结构建模,实现了三种多结构域蛋白质——钙调蛋白、hnRNP A1和hnRNP D0在细胞内的动态结构表征。此外,针对IDP,研究团队提出了两种互补的结构表征策略:一是将XL-MS信息直接转换为距离约束用于IDP的结构计算,二是首先使用全原子分子动力学模拟进行无偏采样,然后基于XL-MS数据对采样结构进行评估和筛选。利用这两种策略,研究团队解码了高迁移率组蛋白HMG-I/Y和HMG-17在细胞内的动态系综构象。
上述成果以“Decoding Protein Dynamics in Cells Using Chemical Cross-Linking and Hierarchical Analysis”为题,于近日发表在《德国应用化学》(Angewandte Chemie International Edition)。该工作的第一作者是1810组博士研究生张蓓蓉。该工作得到了国家重点研发计划、国家自然科学基金、中国科学院青促会等项目的资助。(文/图 张蓓蓉、赵群、龚洲)